肿瘤表达差异基因分析有哪些好用的工具?
部分内容参考 小张聊科研
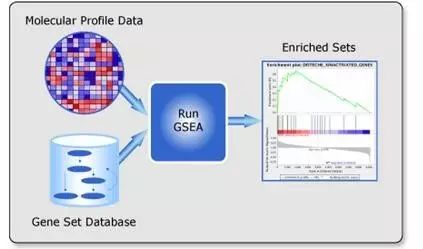
给大家分享一款很好用的带有图形界面的基因富集分析工具GSEA!
【GSEA的用途】
简单来说,GSEA可以帮助你把成百上千万的差异基因结果富集起来,并且将其与明确的生物学通路衔接!在这个过程中,你不需要去指定差异的阈值,一切都会由程序根据具体情况进行调整。
GSEA又叫基因集富集分析 (Gene Set Enrichment Analysis, GSEA),其基本思想是使用预定义的基因集,然后将基因按照在两类样本中的差异表达程度排序,然后检验预定义的基因集合是否在这个排序表的顶部或者底部富集。
GSEA是个图形化操作的软件(对于广大实验狗来说是不是福音?),并且能够给出大量非常专业的分析图表(比如像下面这样):
【GSEA的优势】
一般的差异分析(GO / Pathway)侧重于比较两组间的基因表达差异,集中关注少数几个显著上调或下调的基因。但是有些基因的差异表达虽然不显著,但却有非常重要生物学意义。此外常见的 GO/Pathway 分析也会忽略一些基因的生物特性、基因调控网络之间的关系及基因功能和意义等有价值的信息。
GSEA不需要指定明确的差异基因阈值,算法会根据实际数据的整体趋势, 为研究者们提供了一种合理地解决目前基因表达分析瓶颈问题的方法。
【GSEA富集分析的三步骤】
(1)计算富集分数(Enrichment Score);
(2)估计富集分数的显著性水平;
(3)矫正多重假设检验;
【GSEA的基因集合数据来源】
GSEA的基因集合来源于数据库MSigDB,分为H,C1-C7这几大块。
H: hallmark gene sets (效应)特征基因集合,共50组,如细胞凋亡的特征基因集合、细胞分裂checkpoint的的特征基因集合等。
C1: positional gene sets 位置基因集合,根据染色体位置,共326个。
C2: curated gene sets:(专家)共识基因集合。
C3: motif gene sets:模式基因集合,主要包括microRNA和转录因子靶基因。
C4: computational gene sets:计算基因集合,通过挖掘癌症相关芯片数据定义的基因集合。
C5: GO gene sets:Gene Ontology 基因本体论,包括BP(生物学过程biological process,细胞原件cellular component和分子功能molecular function三部分)。
C6: oncogenic signatures:癌症特征基因集合。
C7: immunologic signatures: 免疫相关基因集合。
【GSEA的下载和安装】
GSEA的下载需要先安装Java,访问http://www.oracle.com/technetwork/java/javase/downloads/jdk8-downloads-2133151.html
根据自己电脑的操作系统下载32位或者64位的Java。
然后访问GSEA的官网:
http://software.broadinstitute.org/gsea/login.jsp下载GSEA(可能需要先注册)。
还想学习更多?想不想学习如何使用GSEA分析自己的数据?
在此安利一下6月2日即将在上海交大医学院开班的第五期《肿瘤生物信息学培训班》,由宇道生物主办,上交大医学院生信中心承办的培训班,讲课老师来自交大医学院、上海生物信息学中心、上海高研院和上海儿童医学中心。
讲课老师均为教授以及一线的生信分析人员,很多讲师自己独立发表过10分以上的生信文章!与其他由公司员工讲课的培训班不同,本培训班主要以教授生物信息学分析技巧和思路为目的,绝对没有推销喔!
亮点如下:
(1)高分经典文章,如NAR,AJHG的文章实例讲解!
(2)小班教学,手把手教分析和作图!
(3)课程经过2016年一整年的磨炼,思路连贯,安排合理!绝不是强行拼凑热点,东一块西一块的“膏药式”课程;)
主办单位:上海宇道生物技术有限公司/上海交通大学医学院医药生物信息学中心
培训地点:上海交通大学医学院(上海市重庆南路280号4号楼103)
本次课程时间:2017年06月02日- 06月04日(报名中)
温馨提示:本次课程包含大量上机操作,请自备笔记本电脑,主办方提供无线网络
本次课程安排:
|
时间 |
授课内容 |
时间 |
|
6月2日 |
肿瘤生物信息学概论 |
8:30 – 9:30 |
|
临床病人肿瘤样本数据库(TCGA/ICGC等)介绍 |
9:45 – 10:45 |
|
| TCGA数据分析方法及经典实例 |
11:00 – 12:00 |
|
|
午餐 |
||
|
TCGA数据应用实例 - 临床病人肿瘤突变位点与蛋白功能的关联分析 |
13:30 – 15:30 |
|
|
上机实践 |
15:45 – 17:45 |
|
|
6月3日 |
肿瘤RNA二代测序数据分析流程与实用软件 |
8:30 – 10:30 |
| R语言分析TCGA/二代测序数据入门 |
10:45 – 11:45 |
|
|
午餐 |
||
|
TCGA数据应用实例-临床病人肿瘤的表达谱分析 |
13:30 – 15:30 |
|
|
上机实践 |
15:45 – 17:45 |
|
|
6月4日 |
二代测序技术在罕见病中的应用实例 |
8:30 – 10:00 |
|
肿瘤生物样本库的建立规范及使用 |
10:15 – 11:45 |
|
|
午餐 |
1
零基础学员通过本课程也能学会基本的肿瘤样本二代测序数据分析方法与常用软件,学会TCGA的数据库的使用,并对如何使用临床病人的肿瘤样本进行研究分析有基本的理解和实践能力。
2
优质讲师团队(上海交通大学医学院、上海生物信息学中心、上海高研院以及多位在一线承担科研工作的研究人员)上机实践及一对一指导
3
小班化教学,每期不超过30人,力求优质高效教学
4
符合实验科学家思维模式的自编全彩教材+装有肿瘤软件和配套上机实践的U盘
报名方式
如何报名参加本次学习班? 请长按下面二维码登记即可!
报名费用
培训费用:2900元(费用包括教材费、培训费、上机费、午餐费)。
优惠政策:截图分享至朋友圈优惠100元/人,3人及以上同时报名,减免200元/人(两种优惠可以叠加使用)。
往届盛况



Pubmed文献助手(显示影响因子、Scihub全文链接、一键翻译摘要等功能!)
如何实现: 长按下面二维码关注公众号:生物医药技术对接平台
实现:
-
Plus工具条:直接显示期刊影响因子、引用次数、参考文献、Scihub全文链接(90%免费全文)等信息。
-
引文索引:可以显示文章参考文献、施引文献。
-
翻译功能:实现中文输入检索式自动翻译为英文;一键翻译摘要。准确度高!
-
搜索过滤系统:精细限定搜索范围。
-
良好的手机端访问体验:支持手机端、电脑端访问。
以蛋白研究之名 揭秘看得见的蛋白互作新技术 Duolink® PLA®
【国家基金委通知】基因信息传递过程中非编码RNA的调控作用机制重大研究计划2017年度项目指南
[王永明教授在线讲座]利用附着体CRISPR技术实现高达100%的编辑效率