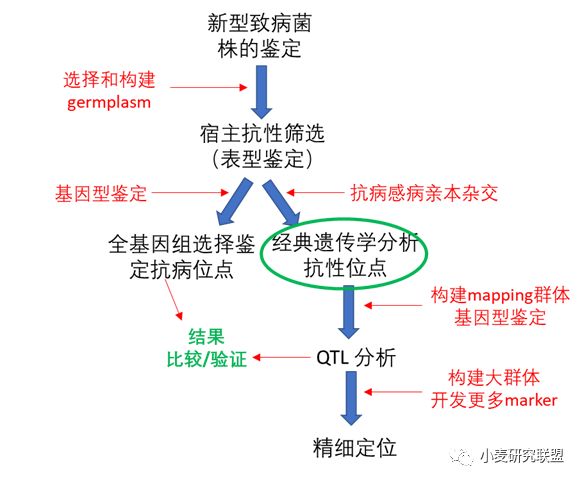
抗病基因鉴定和精细定位
今天借着一篇大麦抗病基因精细定位的文章来聊聊植物抗病那些事~

首先,我做的项目目标之一是鉴定新抗源(identification and characterization of new resistance sources for spot blotch)。所用的植物材料是美国农业部National Small Grains Collection (NSGC) barley collection,总共包括2062份全世界收集来的大麦种质资源(six-rowed大概占60%,two-rowed占40%)。病原菌包括三个不同的致病菌种,其中两个是很早之前鉴定的ND85F和ND90Pr,第三个是新鉴定到的一个菌种ND4008,这个新菌株对目前大规模使用的大麦种质资源都是致病的,所以我们整个研究的novelty和importance就是针对这个新菌株的抗性。接下来小编就结合我博士所在实验室发表的几篇文章来介绍一下这方面的工作。
第一篇:抗性鉴定和遗传分析
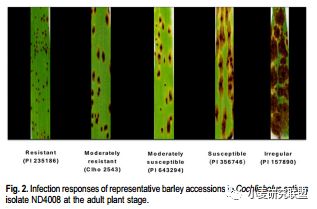
这篇文章主要研究了NSGC 2062个大麦品种对三个不同菌株的表型鉴定,包括所有植株的苗期和部分植株的成熟期抗性。由于是全世界收集来的材料,所以在表型上会出现一些特殊的情况,下图第五个表型就是一种很特别的病理反应,这张图也作为了发表当期杂志的封面图片。
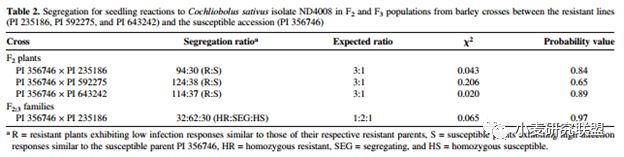
然后我们利用genotype数据把抗病品种做了cluster分析,对鉴定到的抗性资源做了分类。这些数据看起来简单,但仔细分析的话还是可以得到很多有用的结论:比如同样一个群体对一个菌种只有2%抗病,而对于另外一个菌种是只有2%感病,然后对于第三个菌种抗病感病又大概是五五分,这些结果对抗性机制的分析有非常重要的作用。接下来我们又做了Genetics of Spot Blotch Resistance,也就是上边流程图中绿圈的部分。根据我们实验室以及其它课题组的研究,大麦中对先前鉴定的两个菌株的F2表型分离都符合1:3,也就是有显性的感病gene存在。针对新菌株,我们根据抗源的Cluster信息选了几个不同的抗病亲本与同一个感病亲本,做了几个杂交组合,最后genetic分析结果符合3:1,是个显性抗病gene,我们还是相当兴奋的,毕竟跟另外的另外另个菌种的反应是完全不同的两个类型。
题外话:其实表型的分布有很大信息量的,尤其对于有主效基因的性状和群体来说,看一下表型分布,再做个卡方测验,就可以知道或预期QTL analysis要找到几个位点。大家好像都已经习惯把所有表型分布都看成是正态分布,现在也比较少有文章会在讨论的时候把QTL结果跟表型对一下,小编认为这点还蛮重要的。
第二篇:GWAS 分析
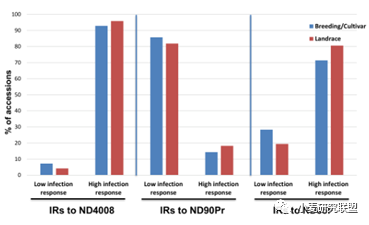
首先在表型数据方面,虽然已经有了前一篇文章,我们还是结合GWAS文章的主题深入挖掘了一些其它结果,比如把表型数据根据barley群体中最大的structure (row-type) 分类描述。然后也对breeding lines/cultivars VS. landraces的表型做了对比,这点虽然跟GWAS的主题没有太大关系,但reviewer对这个图非常喜欢,让我们把它从Supplemental Table 放到了正文中。
对于GWAS方法上的介绍,小编在以前的推送中有比较多的提及QTL mapping 思路总结(二)GWAS-1;GWAS思路总结-2,有兴趣的小伙伴可以查看。
对于结果部分,有两点值的介绍一下:
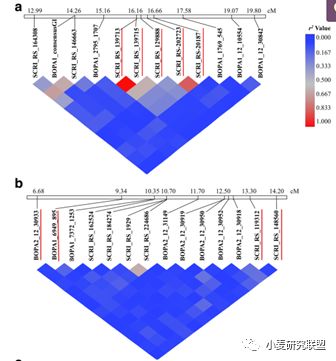
1.对于新菌株,只有一个抗性位点在三个GWAS panel(Whole,Two-row,and Six-row Panel)中都鉴定出来(下面a图)。这个位点包含五个marker(红线标注),但只有两个marker (遗传距离0.1cM) 中在三个panel中都可以鉴定出来,而且这两个marker的p values也是最低的。这些结果被接下来的fine mapping很好的验证,具体比较见第三篇文章介绍。
2. 下面a图中抗性位点(对新菌株的抗性)包含的几个marker的LD已经比较大,而b图中抗性位点(是对另外一个菌株的感病gene)却是处在一个交换热区(红线标注的为significant marker),14个marker分布在不到8cM之内,但任意两个marker之间的LD都小于0.2。由于大麦的genome也已经完全释放,现在结合marker的物理位置和距离,两个图中的LD信息对于未来gene的克隆和验证是有很大帮助的。
题外话:如果是刚接触GWAS,建议还是多读一些以前的文章,那时的文章会有比较详细的方法描述,比如analysis model的选择,背景LD的计算,还有QQ lot背后的数据MSD values等等,现在的文章都不用在present这些结果了。
第三篇:QTL analysis和精细定位
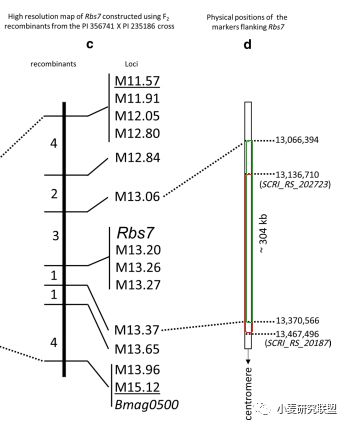
所用的材料就是第一篇文章genetic study中所用到的一个杂交组合。从第一篇文章中我们已经推测出这是一个显性抗病gene,通过GWAS也大概知道了该gene的位置。所以我们首先利用一个200多份材料的F2群体用BSA(SSR marker)的方法对全基因组扫描了一下,确定只有6H(GWAS结果)上有一个紧密连锁位点。然后再利用这个F2群体和用其构建的RIL群体设计更多的SSR标记和CAPS标记来初步定位这个gene。接下来就是用大群体和高密度的marker来进行fine mapping。其实思路和方法都非常简单,尤其大麦spot blotch这个病的表型跟其它病害比起来要简单很多,而且我们做的杂交组合的亲本挑选的又是那种极端类型。
值得一提的是fine mapping和GWAS结果的比较:下图中红色方框为GWAS中最显著的两个marker所在区间(336 kbp),绿色方框是 fine mapping鉴定到的大约304 kbp区间,可以看出二者很好的相互验证。这个例子证明起码在大麦中,GWAS的方法是可以得到一些很好的结果的。而且考虑到大麦是比小麦更严格的自花授粉,理论上来说小麦中如果marker足够密集,用GWAS应该是能直接检测到一些强效应位点的。
这篇文章的另一个重点就是预测了抗病gene可能的抗性机制,关于这点小编下次结合我们在病原菌方面的研究跟大家再介绍一下。
参考文献
Title: Sources and Genetics of Spot Blotch Resistance to a New Pathotype of Cochliobolus sativus in the USDA National Small Grains Collection
Plant Disease 2016
Authors: Yueqiang Leng*, Rui Wang*, Shaukat Ali, Mingxia Zhao, and Shaobin Zhong
Title: Genome-wide association mapping of spot blotch resistance to three different pathotypes of Cochliobolus sativus in the USDA barley core collection
Molecular Breeding 2017
Authors: R Wang, Y Leng, S Ali, M Wang, S Zhong
Title: The gene conferring susceptibility to spot blotch caused by Cochliobolus sativus is located at the Mla locus in barley cultivar Bowman
Theoretical and Applied Genetics 2018
Authors: Yueqiang Leng, Mingxia Zhao, Rui Wang, Brian J Steffenson, Robert S Brueggeman, Shaobin Zhong
Title: Fine mapping of a dominant gene conferring resistance to spot blotch caused by a new pathotype of Bipolaris sorokiniana in barley
Theoretical and Applied Genetics 2018
Authors: R Wang*, Y Leng*, M Zhao*, S Zhong